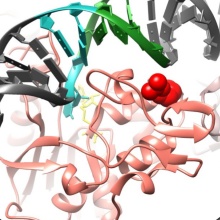
Bis zu 25 Prozent von Patienten mit akuter myeloischer Leukämie (AML) weisen Mutationen in einem Enzym auf, das an der Regulation der DNA-Methylierung beteiligt ist. Forschende um Prof. Albert Jeltsch von der Universität Stuttgart versuchen nun, die biologischen Auswirkungen dieser Mutationen zu verstehen. Gelingt dies, könnte das neue Ansätze zur Behandlung solcher Tumoren aufweisen. Das Projekt wurde von der Wilhelm Sander-Stiftung gefördert.
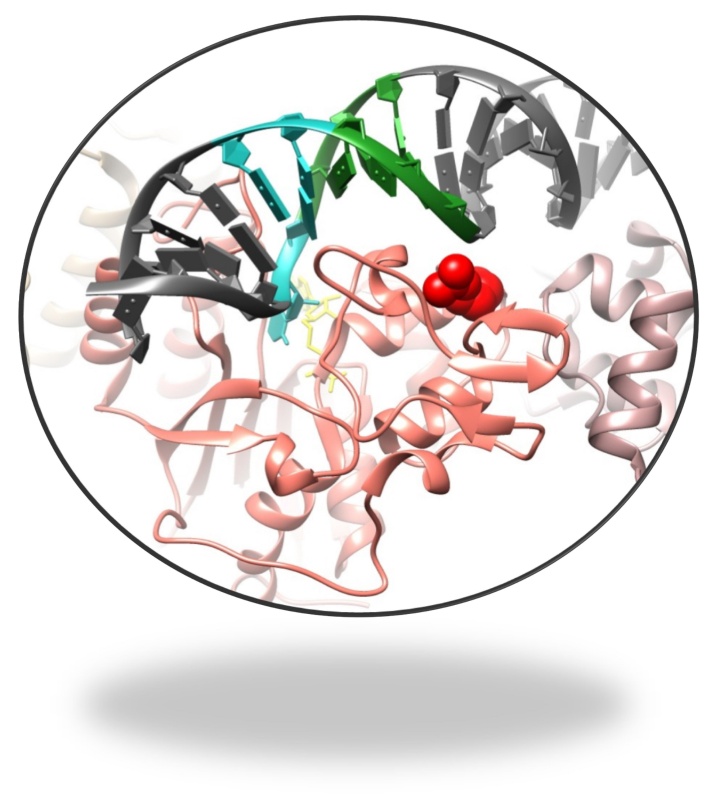
Die Leukämie allgemein ist eine bösartige Erkrankung der Organe, die für die Bildung der roten und weißen Blutkörperchen verantwortlich sind (u.a. Knochenmark und Lymphsystem). Bei Leukämieerkrankten ist der normale Ablauf der Zellproduktion gestört: Die Zellen teilen sich unkontrolliert und ungehemmt, sie reifen nicht aus und können ihre eigentliche Funktion nicht übernehmen. Es wird zwischen akuten Leukämieformen, die schnell und heftig auftreten, und chronischen, die sich langsam entwickeln, unterschieden. Die akute myeloische Leukämie (AML) ist die häufigste akute Leukämieart bei Erwachsenen. Bei der AML entarten unreife Vorstufen von weißen Blutkörperchen, die Granulozyten oder Monozyten.
Ob ein bestimmtes Gen in der Erbsubstanz (DNA) einer Zelle aktiv wird – in der Fachsprache „Genexpression“ – wird von Enzymen gesteuert. Dazu gehören solche, die bestimmte Stellen der DNA verändern, indem sie eine Methylgruppe (chemisch: CH₃), übertragen. Man bezeichnet sie daher als DNA-Methyltransferasen. Die Methylgruppen werden an Stellen der DNA eingefügt, die aus der Abfolge der Basen Cytosin und Guanosin bestehen (CpG-Sequenzen). Sie spielen eine wichtige Rolle bei der Regulation der Genexpression.
In AML-Tumoren findet man oft Veränderungen der DNMT3A-DNA-Methyltransferase. Forscher um Albert Jeltsch haben untersucht, wie diese Mutationen die Eigenschaften des Enzyms verändern. Die beiden häufigsten Varianten sind R882H und R736H. Mithilfe einer neu entwickelten „Deep-Enzymology-Technologie“ (Jeltsch et al., 2021) konnte die Forschungsgruppe zeigen, dass die Variante R882H im Vergleich zum nicht mutierten Enzym (Wildtyp) stark veränderte Methylierungspräferenzen aufweist (Emperle et al., 2019). Die dadurch veränderte Methylierung geht mit einer Fehlregulation mehrere Gene einher, die in enger Verbindung zum Tumorgeschehen stehen. Diese Beobachtungen geben detaillierten Einblick in die Mechanismen, über die die Krebsmutation R882H die Aktivität der Zellen beeinflusst. Da alle Gene in einer Zelle doppelt angelegt sind, kommen unverändertes Enzym (Wildtyp) und R882H-Mutation nebeneinander vor. Es war daher unklar, warum der Effekte der Mutante so stark ausgeprägt ist.
Auch diese Frage konnte in dem durch die Wilhelm Sander-Stiftung geförderten Projekt beantwortet werden (Mack et al., 2022): Die Komplexausbildung von Enzymen aus Wildtyp und R882H-Mutante erfolgt in einer Weise, in der die veränderten Eigenschaften von R882H die des Wildtyp- Enzyms dominieren. Wie R736H, die zweithäufigste Mutation der DNMT3A-DNA-Methyltransferase, auf das Krebsgeschehen wirkt, war zu Beginn des Projekts nicht bekannt. Experimentelle Studien konnten nun zeigen, dass diese Mutante unter bestimmten Bedingungen stimuliert wird (Kunert et al., 2022). Wie bei R882H kann sich dadurch die Methylierung der DNA in den Krebszellen verändern und damit die Genexpression modulieren. Die Hemmung der mutierten DNMT3A-Enzyme R882H und R736H könnte daher zur Behandlung solcher Tumoren beitragen. Quelle: Wilhelm Sander-Stiftung
Über die Wilhelm Sander-Stiftung
Die Wilhelm Sander-Stiftung hat das Forschungsprojekt mit insgesamt knapp 240.000 Euro über 24 Monate unterstützt. Stiftungszweck ist die Förderung der medizinischen Forschung, insbesondere von Projekten im Rahmen der Krebsbekämpfung. Seit Gründung der Stiftung wurden insgesamt über 270 Millionen Euro für die Forschungsförderung in Deutschland und der Schweiz ausbezahlt. Damit ist die Wilhelm Sander-Stiftung eine der bedeutendsten privaten Forschungsstiftungen im deutschen Raum. Sie ging aus dem Nachlass des gleichnamigen Unternehmers hervor, der 1973 verstorben ist.
Fachlicher Kontakt:
Prof. Dr. Albert Jeltsch, Universität Stuttgart, Institut für Biochemie und Technische Biochemie, Abteilung Biochemie, Tel. +49 711 685 64390, E-Mail
Publikationen:
Kunert S, Emperle M, Adam S, Dukatz M, Rajavelu A, Jeltsch A. The R736H cancer mutation in DNMT3A modulates the properties of the FF subunit interface. Biochimie (2022)
Mack A, Emperle M, Schnee P, Adam S, Pleiss J, Bashtrykov P, Jeltsch A. Preferential self-interaction of DNA methyltransferase DNMT3A subunits containing the R882H cancer mutation leads to dominant changes of flanking sequence preferences. J Mol Biol 434, 167482 (2022).
Jeltsch A, Adam S, Dukatz M, Emperle M, Bashtrykov P. Deep enzymology studies
on DNA methyltransferases reveal novel connections between flanking sequences
and enzyme activity. J Mol Biol 433, 167186 (2021).
Emperle M, Adam S, Kunert S, Dukatz M, Baude A, Plass C, Rathert P, Bashtrykov P,
Jeltsch A. Mutations of R882 change flanking sequence preferences of the DNA
methyltransferase DNMT3A and cellular methylation patterns. Nucleic Acids Res 47,
11355-11367 (2019).